UPGMA (unweighted pair group method with arithmetic mean)是一種相對簡單的層次聚類方法。這個方法存在另一種變體 WPGMA。這個方法的創始人被認為是Sokal和Michener 。 [1]
UPGMA 演法構建出一棵有根樹(樹狀圖)表現相似矩陣或相異矩陣中的特徵與結構。在算法裡的每一步,距離最近的兩個集群(子樹)將被組合成一個更高級別的集群。任意兩個集群 和
和 之間的距離,是由所有
之間的距離,是由所有 裡的
裡的 元素和所有
元素和所有 裡的
裡的 元素的距離
元素的距離 的平均值,即每個集群的元素之間的平均距離,其中
的平均值,即每個集群的元素之間的平均距離,其中  和
和 是該兩個集群的基數(集合大小):
是該兩個集群的基數(集合大小):

換句話說,在每一次組合成新集群的步驟中,可以由 和
和 的加權平均給出集群
的加權平均給出集群 和一個新集群
和一個新集群 之間的距離:
之間的距離:

UPGMA 演算法生成的有根樹狀圖是一個超度量樹,該樹需要套用等速率的假設,也就是說根到每個分支尖端的距離皆相等。當尖端是同時採樣的分子數據(即DNA 、 RNA和蛋白質)時,超度量假設就等同於分子鐘假設。
這個示例是基於JC69基因距離矩陣,該矩陣是根據五種細菌的5S 核糖體 RNA序列計算出來的,五種細菌如下所列[2] [3]:
枯草桿菌 Bacillus subtilis(  )
)
嗜熱脂肪芽孢桿菌 <i>Bacillus stearothermophilus</i>(  )
)
魏斯氏菌 Lactobacillus viridescens(  )
)
無原枯草桿菌 Acholeplasma modicum(  )
)
藤黃微球菌 <i>Micrococcus luteus</i>(  )
)
假設有五個物件 和他們之間的相異矩陣
和他們之間的相異矩陣 :
:
|
|
a
|
b
|
c
|
d
|
e
|
| a
|
0
|
17
|
21
|
31
|
23
|
| b
|
17
|
0
|
30
|
34
|
21
|
| c
|
21
|
30
|
0
|
28
|
39
|
| d
|
31
|
34
|
28
|
0
|
43
|
| e
|
23
|
21
|
39
|
43
|
0
|
在這裡, 是最小值,所以將
是最小值,所以將 和
和 集群。
集群。
令 表示現在
表示現在 和
和  的祖先。為了讓
的祖先。為了讓 和
和  與
與  等距,假設
等距,假設 ,這對應到了超度量的假設。在這個範例中:
,這對應到了超度量的假設。在這個範例中:
然後將 更新成一個新的距離矩陣
更新成一個新的距離矩陣 (計算在下方),由於
(計算在下方),由於 和
和 的集群,該矩陣的尺寸減少了一行一列。(
的集群,該矩陣的尺寸減少了一行一列。( 中粗體表示的值是由加權平均計算出的新距離)
中粗體表示的值是由加權平均計算出的新距離)



 中的斜體值不受矩陣更新影響,因為他們與第一個集群中的元素完全美有關連。
中的斜體值不受矩陣更新影響,因為他們與第一個集群中的元素完全美有關連。
現在重複前面的三個步驟,並從新的相異矩陣 開始
開始
|
|
(a,b)
|
c
|
d
|
e
|
| (a,b)
|
0
|
25.5
|
32.5
|
22
|
| c
|
25.5
|
0
|
28
|
39
|
| d
|
32.5
|
28
|
0
|
43
|
| e
|
22
|
39
|
43
|
0
|
在這個矩陣中,  是
是 中的最小值,所以將
中的最小值,所以將 和元素
和元素 集成新群。
集成新群。
令 表示節點
表示節點 和
和 的祖先。由超度量假設可以得到
的祖先。由超度量假設可以得到 三頂點到
三頂點到 的距離相等,即:
的距離相等,即: ,從而可以計算出
,從而可以計算出 到
到 的距離
的距離
然後將 更新成新的距離矩陣
更新成新的距離矩陣 ,數值計算如下:
,數值計算如下:


重複上述動作可以得到 是
是
|
|
((a,b),e)
|
c
|
d
|
| ((a,b),e)
|
0
|
30
|
36
|
| c
|
30
|
0
|
28
|
| d
|
36
|
28
|
0
|
 是:
是:
|
|
((a,b),e)
|
(c,d)
|
| ((a,b),e)
|
0
|
33
|
| (c,d)
|
33
|
0
|
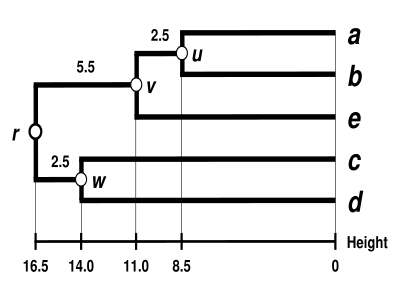
這裡顯示了完成的樹狀圖。[4]它是超度量的,所有尖端(  到
到 ) 與
) 與 等距離 :
等距離 :

這個樹狀圖的根是它最深的節點 。
。
構建 UPGMA 樹的算法有 時間複雜度。使用一個堆維護兩個即群之間的距離可以使時間達到
時間複雜度。使用一個堆維護兩個即群之間的距離可以使時間達到 . 另外Fionn Murtagh 提出了一個
. 另外Fionn Murtagh 提出了一個 時空複雜度的算法。 [5]
時空複雜度的算法。 [5]
- ^ Sokal, Michener. A statistical method for evaluating systematic relationships. University of Kansas Science Bulletin. 1958, 38: 1409–1438.
- ^ Erdmann VA, Wolters J. Collection of published 5S, 5.8S and 4.5S ribosomal RNA sequences. Nucleic Acids Research. 1986,. 14 Suppl (Suppl): r1–59. PMC 341310
 . PMID 2422630. doi:10.1093/nar/14.suppl.r1.
. PMID 2422630. doi:10.1093/nar/14.suppl.r1.
- ^ Olsen GJ. Phylogenetic analysis using ribosomal RNA. Methods in Enzymology. 1988, 164: 793–812. PMID 3241556. doi:10.1016/s0076-6879(88)64084-5.
- ^ Swofford DL, Olsen GJ, Waddell PJ, Hillis DM. Hillis , 編. Phylogenetic inference. Sunderland, MA: Sinauer. 1996: 407–514. ISBN 9780878932825.
- ^ Murtagh F. Complexities of Hierarchic Clustering Algorithms: the state of the art. Computational Statistics Quarterly. 1984, 1: 101–113.

 . PMID 2422630. doi:10.1093/nar/14.suppl.r1.
. PMID 2422630. doi:10.1093/nar/14.suppl.r1.











































