代谢物组
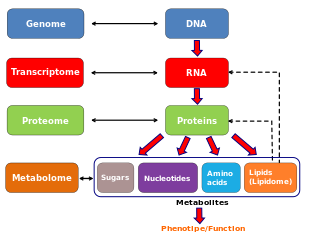
代谢物组(英語:Metabolome)是指在一个生物样品中发现的完整的一套小分子化学物质。[1]所述生物样品可以是一个细胞,一个细胞器,一个器官,一个组织,一个组织提取物,一个生物流体或整个生物体。在给定的代谢物组发现的小分子的化学物质可能既包括内源性代谢物,它们是由生物体天然产生的(如氨基酸,有机酸,核酸,脂肪酸,胺,糖,维生素,辅因子,颜料,抗生素等),又包括外源化学物质(如药物,环境污染物,食品添加剂,毒素和其它生物异源物质),它们不是由生物体天然产生的。[2][3]换句话说,代谢物组既有内源性代谢物组又有外源性代谢物组。内源性代谢物组可进一步细分为包括“初级”的和“次级”的代谢物组(特别是指植物的或微生物的代谢物组时)。初级代谢产物是直接参与正常的生长,发育和繁殖。次级代谢产物并不直接参与这些过程,但通常具有重要的生态功能。次级代谢产物可能包括色素,抗生素或部分生物异源物质代谢废物的衍生产品。一个小分子有资格作为一种代谢物,或者被认为是代谢物组的一部分,必须通常具有分子量<1500道尔顿。[2]这意味着例如糖脂,多糖,短肽(<14个氨基酸)和小的寡核苷酸(<5个碱基)的分子可以视为代谢物质或代谢物组的成分。另一方面,非常大的高分子,如蛋白质,信使RNA,核糖体RNA,microRNA和脱氧核糖核酸(DNA)绝对不是代谢物质,并且不被认为是代谢物组的一部分。代谢物组的研究被称为代谢物组学(英語:Metabolomics)。
起源
[编辑]代谢物组(Metabolome)的这个英文词看起来是“代谢物(metabolite)”和“染色体(chromosome)”这个英文词的混合。它被构建为暗示代谢物由基因间接编码或作用于基因和基因产物。术语“代谢物组”(英語:Metabolome)首先在1998年[1][4]被使用,并且可能是与现有的生物术语相匹配的,指的是完整的基因集合(基因组),完整的蛋白质集合(蛋白质组)和完整的转录物集合(转录物组)。关于代谢物组的第一本书于2003年出版[5]。第一本致力于代谢物组学的杂志(简称《代谢物组学》)于2005年推出,目前由Royston Goodacre博士主编。 关于代谢组学分析的一些更重要的早期论文列在下面的参考文献中[6][7][8][9]。
测量代谢物组
[编辑]代谢物组反映了生物体的基因组与其环境之间的相互作用。结果,生物体的代谢物组可以作为其表型的优良探针(即其基因型及其环境的产物)。可以使用许多不同的技术包括核磁共振波谱法(NMR)和质谱法(MS)来测量(识别,定量,或分类)代谢产物。大多数质谱(MS)方法必须连接到各种形式的液相色谱(LC),气相色谱(GC)或毛细管电泳(CE)以促进化合物分离。每种方法通常能够一次识别或表征50-5000种不同的代谢物或代谢物“特征”,这取决于所使用的仪器或方案。参见图2,了解不同分析方法与其灵敏度之间的关系。目前,不可能通过单一的分析方法分析代谢物组的整个范围。
与基因组甚至蛋白质组不同,代谢物组是一个高度动态的实体,可以在几秒钟或几分钟的时间内发生剧烈变化。因此,使用核磁共振波谱法(NMR)和质谱法(MS)的代谢组学的修改版本,在多个时间段或短时间间隔内测量代谢物的兴趣越来越大。
代谢物组数据库
[编辑]因为生物体的代谢组织主要由其基因组定义,不同的物种将具有不同的代谢物组。事实上,番茄的代谢物组与苹果的代谢物组不同之处在于这两种水果味道如此不同。此外,与这些器官和组织相关的不同组织,不同器官和生物流体也可以具有明显不同的代谢物组。不同生物体和不同组织/生物流体具有这样不同的代谢物组的事实已经导致生物特异性生物流体特异性代谢物组数据库的发展。一些知名的代谢组学数据库包括人类代谢物组数据库或HMDB[10],酵母代谢组数据库或YMDB[11],大肠杆菌代谢物组数据库或ECMDB[12],拟南芥代谢物组数据库或AraCyc[13],以及尿代谢物组数据库[14],脑脊液(CSF)代谢物组数据库[15],和血清代谢物组学数据库[16] 。后三个数据库是人类生物流体特有的。还存在许多非常流行的一般代谢物数据库,包括京都基因与基因组百科全书(KEGG)[17],MetaboLights[18],戈尔姆代谢物组数据库[19],MetaCyc[20],LipidMaps[21],和Metlin[22]。代谢物组数据库与代谢物数据库可以进行区分,因为代谢物数据库包含来自多个生物体的轻微注释的或天然的代谢物数据,而代谢组学数据库包含特定生物体的丰富详细的和大量参考的化学的,途径的,光谱的和代谢物浓度数据。
参看
[编辑]参考资料
[编辑]- ^ 1.0 1.1 Oliver, SG; Winson MK; Kell DB; Baganz F. Systematic functional analysis of the yeast genome. Trends Biotechnol. September 1998, 16 (9): 373–8. PMID 9744112. doi:10.1016/S0167-7799(98)01214-1.
- ^ 2.0 2.1 Wishart, DS. Current progress in computational metabolomics. Brief Bioinform. September 2007, 8 (5): 279–93. PMID 17626065. doi:10.1093/bib/bbm030.
- ^ Nordström, A; O'Maille G; Qin C; Siuzdak G. Nonlinear data alignment for UPLC-MS and HPLC-MS based metabolomics: quantitative analysis of endogenous and exogenous metabolites in human serum. Anal. Chem. 2006, 78 (10): 3289–95. PMID 16689529. doi:10.1021/ac060245f.
- ^ Tweeddale, H; Notley-McRobb L; Ferenci T. Effect of slow growth on metabolism of Escherichia coli, as revealed by global metabolite pool ("metabolome") analysis.. J Bacteriol. Oct 1998, 180 (19): 5109–16. PMC 107546
 . PMID 9748443.
. PMID 9748443.
- ^ Harrigan, G. G. & Goodacre, R. (eds). Metabolic Profiling: Its Role in Biomarker Discovery and Gene Function Analysis.. Boston: Kluwer Academic Publishers. 2003. ISBN 1-4020-7370-4.
- ^ Fiehn, O.; Kloska S; Altmann T. Integrated studies on plant biology using multiparallel techniques.. Curr. Opin. Biotechnol. 2001, 12 (1): 82–6. PMID 11167078. doi:10.1016/S0958-1669(00)00165-8.
- ^ Fiehn, O. Combining Genomics, Metabolome Analysis, and Biochemical Modelling to Understand Metabolic Networks.. Comp. Funct. Genomics. 2001, 2 (3): 155–68. PMC 2447208
 . PMID 18628911. doi:10.1002/cfg.82.
. PMID 18628911. doi:10.1002/cfg.82.
- ^ Weckwerth, W. Metabolomics in systems biology. Annu Rev Plant Biol. 2003, 54: 669–89. PMID 14503007. doi:10.1146/annurev.arplant.54.031902.135014.
- ^ Goodacre, R.; Vaidyanathan S; Dunn WB; Harrigan GG; Kell DB. Metabolomics by numbers: acquiring and understanding global metabolite data.. Trends Biotechnol. 2004, 22 (5): 245–52. PMID 15109811. doi:10.1016/j.tibtech.2004.03.007.
- ^ Wishart, DS; Tzur D; Knox C; et al. HMDB: the Human Metabolome Database. Nucleic Acids Research. January 2007, 35 (Database issue): D521–6. PMC 1899095
 . PMID 17202168. doi:10.1093/nar/gkl923.
. PMID 17202168. doi:10.1093/nar/gkl923.
- ^ Jewison T, Neveu V, Lee J, Knox C, Liu P, Mandal R, Murthy RK, Sinelnikov I, Guo AC, Wilson M, Djoumbou Y, Wishart DS. The Yeast Metabolome Database.. Nucleic Acids Research. Jan 2012, 40 (Database issue): D815–20. PMC 3245085
 . PMID 22064855. doi:10.1093/nar/gkr916.
. PMID 22064855. doi:10.1093/nar/gkr916.
- ^ Guo, A.; Jewison T; Wilson M; Liu Y; Knox C; Djoumbou Y; Lo P; Mandal R; Krishnamurthy R; Wishart DS. ECMDB: the E. coli Metabolome Database. Nucleic Acids Res. Jan 2013, 41 (Database issue): D625–30. PMC 3531117
 . PMID 23109553. doi:10.1093/nar/gks992.
. PMID 23109553. doi:10.1093/nar/gks992.
- ^ Mueller, LA; Zhang P; Rhee SY. AraCyc: a biochemical pathway database for Arabidopsis.. Plant Physiol. June 2003, 132 (2): 453–60. PMC 166988
 . PMID 12805578. doi:10.1104/pp.102.017236.
. PMID 12805578. doi:10.1104/pp.102.017236.
- ^ Bouatra S, Aziat F, Mandal R, Guo AC, Wilson MR, Knox C, Bjorndahl TC, Krishnamurthy R, Saleem F, Liu P, Dame ZT, Poelzer J, Huynh J, Yallou FS, Psychogios N, Dong E, Bogumil R, Roehring C, Wishart DS. The human urine metabolome.. PLoS ONE. Sep 2013, 8 (9): e73076. PMC 3762851
 . PMID 24023812. doi:10.1371/journal.pone.0073076.
. PMID 24023812. doi:10.1371/journal.pone.0073076.
- ^ Mandal, R; Guo AC; Chaudhary KK; Liu P; Yallou FS; Dong E; Aziat F; Wishart DS. Multi-platform characterization of the human cerebrospinal fluid metabolome: a comprehensive and quantitative update. Genome Med. Apr 2012, 4 (4): 38. PMC 3446266
 . PMID 22546835. doi:10.1186/gm337.
. PMID 22546835. doi:10.1186/gm337.
- ^ Psychogios N, Hau DD, Peng J, Guo AC, Mandal R, Bouatra S, Sinelnikov I, Krishnamurthy R, Eisner R, Gautam B, Young N, Xia J, Knox C, Dong E, Huang P, Hollander Z, Pedersen TL, Smith SR, Bamforth F, Greiner R, McManus B, Newman JW, Goodfriend T, Wishart DS. The human serum metabolome.. PLoS ONE. Feb 2011, 6 (2): e16957. PMC 3040193
 . PMID 21359215. doi:10.1371/journal.pone.0016957.
. PMID 21359215. doi:10.1371/journal.pone.0016957.
- ^ Kanehisa, M.; Goto S. KEGG: Kyoto Encyclopedia of Genes and Genomes. Nucleic Acids Res. 2000, 28 (1): 27–30. PMC 102409
 . PMID 10592173. doi:10.1093/nar/28.1.27.
. PMID 10592173. doi:10.1093/nar/28.1.27.
- ^ Haug K, Salek RM, Conesa P, Hastings J, de Matos P, Rijnbeek M, Mahendraker T, Williams M, Neumann S, Rocca-Serra P, Maguire E, González-Beltrán A, Sansone SA, Griffin JL, Steinbeck C. MetaboLights--an open-access general-purpose repository for metabolomics studies and associated meta-data. Nucleic Acids Res. 2013, 41 (Database issue): D781–6. PMC 3531110
 . PMID 23109552. doi:10.1093/nar/gks1004.
. PMID 23109552. doi:10.1093/nar/gks1004.
- ^ Kopka, J; Schauer N; Krueger S; Birkemeyer C; Usadel B; Bergmuller E; Dormann P; Weckwerth W; Gibon Y; Stitt M; Willmitzer L; Fernie AR; Steinhauser D. GMD@CSB.DB: the Golm Metabolome Database. Bioinformatics. 2005, 21 (8): 1635–1638. PMID 15613389. doi:10.1093/bioinformatics/bti236.
- ^ Caspi R, Altman T, Dale JM, Dreher K, Fulcher CA, Gilham F, Kaipa P, Karthikeyan AS, Kothari A, Krummenacker M, Latendresse M, Mueller LA, Paley S, Popescu L, Pujar A, Shearer AG, Zhang P, Karp PD. The MetaCyc database of metabolic pathways and enzymes and the BioCyc collection of pathway/genome databases. Nucleic Acids Res. January 2010, 38 (Database issue): D473–9. PMC 2808959
 . PMID 19850718. doi:10.1093/nar/gkp875.
. PMID 19850718. doi:10.1093/nar/gkp875.
- ^ Fahy, E.; Sud M; Cotter D; Subramaniam S. LIPID MAPS online tools for lipid research.. Nucleic Acids Res. July 2007, 35 (Web Server issue): W606–12. PMC 1933166
 . PMID 17584797. doi:10.1093/nar/gkm324.
. PMID 17584797. doi:10.1093/nar/gkm324.
- ^ Smith, CA; I'Maille G; Want EJ; Qin C; Trauger SA; Brandon TR; Custodio DE; Abagyan R; Siuzdak G. METLIN: a metabolite mass spectral database.. Ther Drug Monit. 2005, 27 (6): 747–51. PMID 16404815. doi:10.1097/01.ftd.0000179845.53213.39.
